สารประกอบลิกแนนและยีน Pinoresinol-lariciresinol Reductases 3 ในข้าวพันธุ์ไรซ์เบอรี่
DOI:
https://doi.org/10.55164/jgrdi.v1i1.646คำสำคัญ:
ลิกแนน, ข้าวไรซ์เบอรี่, เอนไซม์ Pinoresinol-lariciresinol Reductase (PLR), ยีน OsPLR3บทคัดย่อ
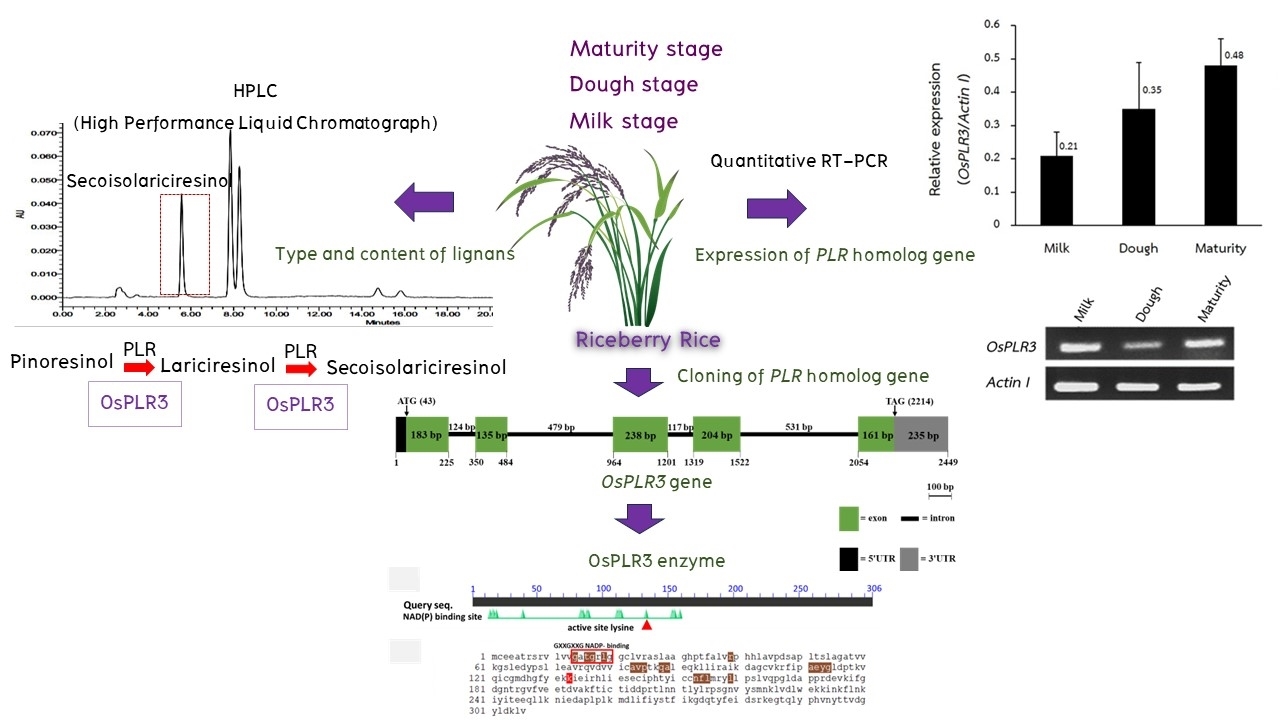
วัตถุประสงค์ของงานวิจัยในครั้งนี้ คือ เพื่อตรวจสอบชนิดและปริมาณสารลิกแนนในเมล็ดข้าวไรซ์เบอรี่ โดยเทคนิค HPLC (High Performance Liquid Chromatography) และโคลนยีนที่กำหนดเอนไซม์ Pinoresinol-lariciresinol Reductase (PLR) ในวิถีการสังเคราะห์สารลิกแนนในข้าวไรซ์เบอรี่ จากผลการทดลองพบสารลิกแนนชนิดซีโคไอโซลาริซิเรซินอลปริมาณ 1.70 ± 0.20 1.96 ± 0.25 และ 3.14 ± 0.19 มิลลิกรัม/กรัมข้าวบด ในระยะน้ำนม ข้าวเม่า และข้าวสาร ตามลำดับ เมื่อวิเคราะห์โครงสร้างของยีน PLR Homolog จากจีโนมข้าวไรซ์เบอรี่ พบว่า ยีน OsPLR3 มีขนาด 2,449 คู่เบส ประกอบด้วย 5 เอกซอน และ 4 อินทรอน มีบริเวณที่กำหนดรหัสกรดอะมิโน 921 นิวคลีโอไทด์ แปลรหัสได้สายพอลิเพปไทด์ 306 กรดอะมิโน โดยพบโดเมนอนุรักษ์ Rossmann-fold NAD(P)(+)-binding Proteins และกรดอะมิโนไลซีน (K) ทำหน้าเร่งปฏิกิริยาดีไฮโดรจีเนชันของเอนไซม์ PLR จากการวิเคราะห์การแสดงออกของยีน OsPLR3 ในแต่ละระยะของการพัฒนาเมล็ด พบว่ายีนมีการแสดงออกต่ำสุดในระยะน้ำนม แล้วเพิ่มขึ้นในระยะข้าวเม่า สูงสุดในระยะข้าวสาร ซึ่งสอดคล้องกับปริมาณการสะสมสารซีโคไอโซลาริซิเรซินอลในเมล็ด จึงคาดว่าปริมาณการสะสมซีโคไอโซลาริซิเรซินอลในเมล็ดข้าวถูกควบคุมโดยยีน OsPLR3
เอกสารอ้างอิง
Kitts, D. D., Yuan, Y. V., Wijewickreme, A. N., & Thompson, L. U. (1999). Antioxidant activity of the flaxseed lignan secoisolariciresinol diglycoside and its mammalian lignan metabolites enterodiol and enterolactone. Molecular and Cellular Biochemistry, 202(1-2), 91-100. https://doi.org/10.1023/a:1007022329660.
Prasad, K. (2002). Suppression of phosphoenolpyruvate carboxykinase gene expression by secoisolariciresinol diglucoside (SDG), a new antidiabetic agent. International Journal of Angiology, 11, 107-109. https://doi.org/10.1007/BF01616377
Pan, A., Sun, J., Chen, Y., Ye, X., Li, H., Yu, Z., Wang, Y., Gu, W., Zhang, X., Chen, X., Demark-Wahnefried, W., Liu, Y., & Lin, X. (2007). Effects of a flaxseed-derived lignan supplement in type 2 diabetic patients: a randomized, double-blind, cross-over trial. PLOS ONE, 2(11), e1148. https://doi.org/10.1371/journal.pone.0001148
Prasad, K., & Dhar, A. (2016). Flaxseed and Diabetes. Current Pharmaceutical Design, 22(2), 141-144. http://dx.doi.org/10.2174/1381612822666151112151230
Prasad, K. (2000). Oxidative stress as a mechanism of diabetes in diabetic BB prone rats: Effect of secoisolariciresinol diglucoside (SDG). Molecular and Cellular Biochemistry, 209, 89-96. https://doi.org/10.1023/a:1007079802459
Prasad, K., Mantha, S., Muir, A., & Westcott, N. (2000). Protective effect of secoisolariciresinol diglucoside against streptozotocin-induced diabetes and its mechanism. Molecular and Cellular Biochemistry, 206, 141-150. https://doi.org/10.1023/a:1007018030524
Sun, Q., Wedick, N. M., Pan, A., Townsend, M. K., Cassidy, A., Franke, A. A., Rimm, E. B., Hu, F. B., & van Dam, R. M. (2014). Gut microbiota metabolites of dietary lignans and risk of type 2 diabetes: A prospective investigation in two cohorts of U.S. women. Diabetes Care, 37(5), 1287-1295. https://doi.org/10.2337/dc13-2513
Cornwell, T., Cohick, W., & Raskin, I. (2004). Dietary phytoestrogens and health. Phytochemistry, 65(8), 995-1016. https://doi.org/10.1016/j.phytochem.2004.03.005
Simbalista, R. L., Sauerbronn, A. V., Aldrighi, J. M., & Arêas, J. A. G. (2010). Consumption of a flaxseed-rich food is not more effective than a placebo in alleviating the climacteric symptoms of postmenopausal women 1, 2. The Journal of Nutrition, 140(2), 293-297. https://doi.org/10.3945/jn.109.113886
Saleem, M., Kim, H. J., Ali, M. S., & Lee, Y. S. (2005). An Update on Bioactive Plant Lignans. Natural Product Reports, 22, 696-716. https://doi.org/10.1039/b514045p
Hano, C., Martin, I., Fliniaux, O., Legrand, B., Gutierrez, L., Arroo, R. R. J., Mesnard, F., Lamblin, F., & Lainé, E. (2006). Pinoresinol-lariciresinol reductase gene expression and secoisolariciresinol diglucoside accumulation in developing flax (Linum usitatissimum) seeds. Planta, 224, 1291-1301. https://doi.org/10.1007/s00425-006-0308-y
Min, B., Gu, L., McClung, A. M., Bergman, C. J., & Chen, M.-H. (2012). Free and bound total phenolic concentrations, antioxidant capacities, and profiles of proanthocyanidins and anthocyanins in whole grain rice (Oryza sativa L.) of different bran colours. Food Chemistry, 133(3), 715-722. https://doi.org/10.1016/j.foodchem.2012.01.079
Leardkamolkarn, V., Thongthep, W., Suttiarporn, P., Kongkachuichai, R., Wongpornchai, S., & Wanavijitr, A. (2011). Chemopreventive properties of the bran extracted from a newly-developed Thai rice: The Riceberry. Food Chemistry, 125(3), 978-985. https://doi.org/10.1016/j.foodchem.2010.09.093
Hutabarat, L. S., Greenfield, H., & Mulholland, M. (2000). Quantitative determination of isoflavones and coumestrol in soybean by column liquid chromatography. Journal of Chromatography A, 886(1-2), 55–63. https://doi.org/10.1016/s0021-9673(00)00444-1
Kuhnle, G. G. C., Dell'Aquila, C., Low, Y. L., Kussmaul, M., & Bingham, S. A. (2007). Extraction and quantification of phytoestrogens in foods using automated solid-phase extraction and LC/MS/MS. Analytical Chemistry, 79(23), 9234–9239. https://doi.org/10.1021/ac701732r
Saghai-Maroof, M. A., Soliman, K. M., Jorgensen, R. A., & Allard, R. W. (1984). Ribosomal DNA spacer-length polymorphisms in barley: mendelian inheritance, chromosomal location, and population dynamics. Proceedings of the National Academy of Sciences of the United States of America, 81(24), 8014–8018. https://doi.org/10.1073/pnas.81.24.8014
Sambrook, J., Fritsch, E. F., & Maniatis, T. (1989). Molecular Cloning: A Laboratory Manual (2nd ed.). Spring Harbor Laboratory Press.
Altschul, S. F., Gish, W., Miller, W., Myers, E. W., & Lipman, D. J. (1990). Basic local alignment search tool. Journal of Molecular Biology, 215(3), 403-410. https://doi.org/10.1016/S0022-2836(05)80360-2
Kapustin, Y., Souvorov, A., Tatusova, T., & Lipman, D. (2008). Splign: algorithms for computing spliced alignments with identification of paralogs. Biology Direct, 3, 20. https://doi.org/10.1186/1745-6150-3-20
Gasteiger, E., Hoogland, C., Gattiker, A., Duvaud, S., Wilkins, M. R., Appel, R. D. & Bairoch, A. (2005). Protein identification and analysis tools on the ExPASy server. In J.M. Walker (ed.). The Proteomics Protocols Handbook (pp. 571-607). Humana Press. https://doi.org/10.1385/1-59259-890-0:571
Lu, S., Wang, J., Chitsaz, F., Derbyshire, M. K., Geer, R. C., Gonzales, N. R., Gwadz, M., Hurwitz, D. I., Marchler, G. H., Song, J. S., Thanki, N., Yamashita, R. A., Yang, M., Zhang, D., Zheng, C., Lanczycki, C. J., & Marchler-Bauer, A. (2020). CDD/SPARCLE: the conserved domain database in 2020. Nucleic Acids Research, 48(D1), D265–D268. https://doi.org/10.1093/nar/gkz991
Czechowski, T., Stitt, M., Altmann, T., Udvardi, M. K., & Scheible, W. R. (2005). Genome-wide identification and testing of superior reference genes for transcript normalization in Arabidopsis. Plant Physiology, 139(1), 5–17. https://doi.org/10.1104/pp.105.063743
Min, T., Kasahara, H., Bedgar, D. L., Youn, B., Lawrence, P. K., Gang, D. R., Halls, S. C., Park, H., Hilsenbeck, J. L., Davin, L. B., Lewis, N. G., & Kang, C. (2003). Crystal structures of pinoresinol-lariciresinol and phenylcoumaran benzylic ether reductases and their relationship to isoflavone reductases. Journal of Biological Chemistry, 278(50), 50714–50723. https://doi.org/10.1074/jbc.M308493200
Markulin, L., Corbin, C., Renouard, S., Drouet, S., Gutierrez, L., Mateljak, I., Auguin, D., Hano, C., Fuss, E., & Lainé, E. (2019). Pinoresinol-lariciresinol reductases, key to the lignan synthesis in plants. Planta, 249(6), 1695–1714. https://doi.org/10.1007/s00425-019-03137-y
Saad, N., Ismail, N., Mastuki, S. N., Leong, S. W., Chia, S. L., & Abdullah, C. A. C. (2022). Chapter 16 - Rice bran oil main bioactive compounds and biological activities. In A. A. Mariod (ed.), Multiple Biological Activities of Unconventional Seed Oils. (pp. 195-213). Academic Press. https://doi.org/10.1016/B978-0-12-824135-6.00017-9
von Heimendahl, C. B. I., Schäfer, K. M., Eklund, P., Sjöholm, R., Schmidt, T. J., & Fuss, E. (2005). Pinoresinol -lariciresinol reductases with different stereospecificity from Linum album and Linum usitatissimum. Phytochemistry, 66(11), 1254–1263. https://doi.org/10.1016/j.phytochem.2005.04.026
Hemmati, S., von Heimendahl, C. B. I., Klaes, M., Alfermann, A. W., Schmidt, T. J., & Fuss, E. (2010). Pinoresinol - lariciresinol reductases with opposite enantiospecificity determine the enantiomeric composition of lignans in the different organs of Linum usitatissimum L. Planta Medica, 76(9), 928–934. https://doi.org/10.1055/s-0030-1250036
Chiang, N.-T., Ma, L.-T., Lee, Y.-R., Tsao, N.-W., Yang, C. –K., Wang, S.-Y., & Chu, F.-H. (2018). The gene expression and enzymatic activity of pinoresinol-lariciresinol reductase during wood formation in Taiwania cryptomerioides Hayata. Holzforschung, 73(2), 197–208. https://doi.org/10.1515/hf-2018-0026
Fujita, M., Gang, D. R., Davin, L. B., & Lewis, N. G. (1999). Recombinant pinoresinol - lariciresinol reductases from western red cedar (Thuja plicata) catalyze opposite enantiospecific conversions. Journal of Biological Chemistry, 274(2), 618–627. https://doi.org/10.1074/jbc.274.2.618
ดาวน์โหลด
เผยแพร่แล้ว
รูปแบบการอ้างอิง
ฉบับ
ประเภทบทความ
สัญญาอนุญาต
ลิขสิทธิ์ (c) 2023 บัณฑิตวิทยาลัย มหาวิทยาลัยทักษิณ

อนุญาตภายใต้เงื่อนไข Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International License.


